Statistisches
Modell & Auswertung
Specify a model
Nach Beendigung des Preprocessing muss nun das statischtische Modell definiert werden. Eingang in diese
Definition finden Paradigmenparameter wie Interscaninterval.
Anzahl der Bilder pro Messung, Anzahl der Trails,
Anzahl der Bilder je Trail etc..
Anhand des nachfolgendes Beispiel
soll die Erstellung des Modells dargestellt werden:
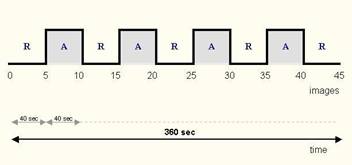
![]() fMRI Messung mit 45 Bildern / Messung
fMRI Messung mit 45 Bildern / Messung
![]() Interscanninterval: 8sec
Interscanninterval: 8sec
![]() Blockdesignparadigma mit 4 ON und 5 OFF, Phasen, alternierend
Blockdesignparadigma mit 4 ON und 5 OFF, Phasen, alternierend
![]() je 5 Bilder
pro ON/OFF Phase
je 5 Bilder
pro ON/OFF Phase
![]() ein Trail,
d.h. nur die ON Phasen werde gewertet, die OFF-Phasen
sind Ruhe Phasen - wenn in der Ruhe (OFF) Handlungen ausgeführt werden (z.B.
Fixieren einer optischen Figur) kann man die OFF-Phase
als eigenen Trail definieren um nacher
die Trails voneinander abziehen zu können
ein Trail,
d.h. nur die ON Phasen werde gewertet, die OFF-Phasen
sind Ruhe Phasen - wenn in der Ruhe (OFF) Handlungen ausgeführt werden (z.B.
Fixieren einer optischen Figur) kann man die OFF-Phase
als eigenen Trail definieren um nacher
die Trails voneinander abziehen zu können
Achtung, die Ergebnisfiles werden in den folgendenden
Schritten nicht automatische zu den bearbeiteten Datensätzen dazugeschrieben,
sondern in das aktuelle Working-directory geschrieben à entsprechendes Directory auswählen!
 fMRI – models anklicken
fMRI – models anklicken
what would you like to do : Specify a model
interscan interval: 8 (Zeit zwischen 2 Scans in sec)
scans per session : 45 (Anzahl der Bilder je Messung)
Nr. of conditions or trail : 1 (Anzahl
der Trails bzw. Testkonditionen)
select number of conditions : trail 1 (Name für Taril z.B. Fingertippen
ON)
stocastic design : no (ist in dem Design vom Zufall abhängig?)
SOA (scans of asynchronie)
: fixed (ist das Design fix bzw. symetrisch
– gleiche Phasen, -längen, -bilderanzahl etc.?)
SOA (scans of as
for trail 1) : 10 (Anzahl der Scans zwischen dem
ersten Beginn von Trail 1bis zum nächsten Beginn des Trails)
Time for first
trail (scan) : 5 (wann beginnt der erste Taril?
– in Anzahl der Scans rechnen, erstes Bild zählt als
0)
Parametric modulation : none
are these trail : epochs (à Block-Design)
select type of response : box car
convolve with hrf : yes (Zeitlich
Ableitung der hämodynamic Response Funktion)
add temporal derivate : no
epoch length : 5 (Anzahl der Bilder je Epoche/Phase)
interaction among trails : no (interagieren
die Trails?)
user specified regressor
: 0
Diagramme erscheinen
SPM_fMRIDesMtx.mat wird im aktuellen Working-directory angelegt
Estimate

fMRI
– models anklicken
what would you like to do: Estimate a specifed
model
Fenster
öffnet sich :
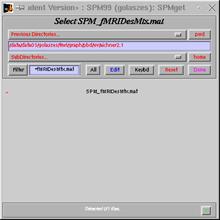
![]() SPM_FMRiDesMtx.mat anklicken
SPM_FMRiDesMtx.mat anklicken ![]() Done
anklicken
Done
anklicken
Fenster
öffnet sich :
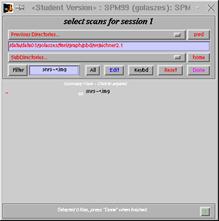
![]() alle snrs-4...img anklicken
alle snrs-4...img anklicken ![]() Done anklicken
Done anklicken
Remove global effects
: no
High pass filter
: specify
Session ... period : 175 (Vorgabe annehmen)
Low pass filter : HRF
Model intrinsic correlation
: none
Setup trial-specific F-contrast : yes
Estimate : now
|
Säule erscheint – warten |
SPM.mat wird im aktuellen Working-directory angelegt
Results
 Results anklicken
Results anklicken
Fenster öffnet
sich :
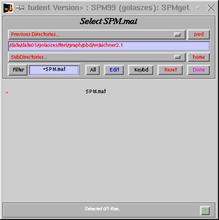
![]() SPM.mat anklicken
SPM.mat anklicken![]() Done anklicken
Done anklicken
Fenster
öffnet sich :

Fenster
öffnet sich :
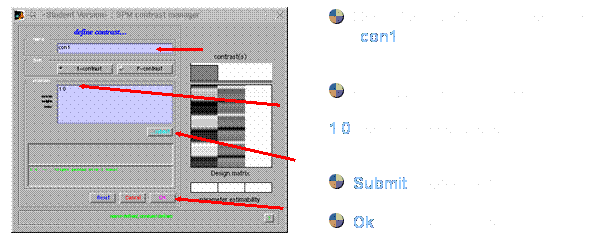
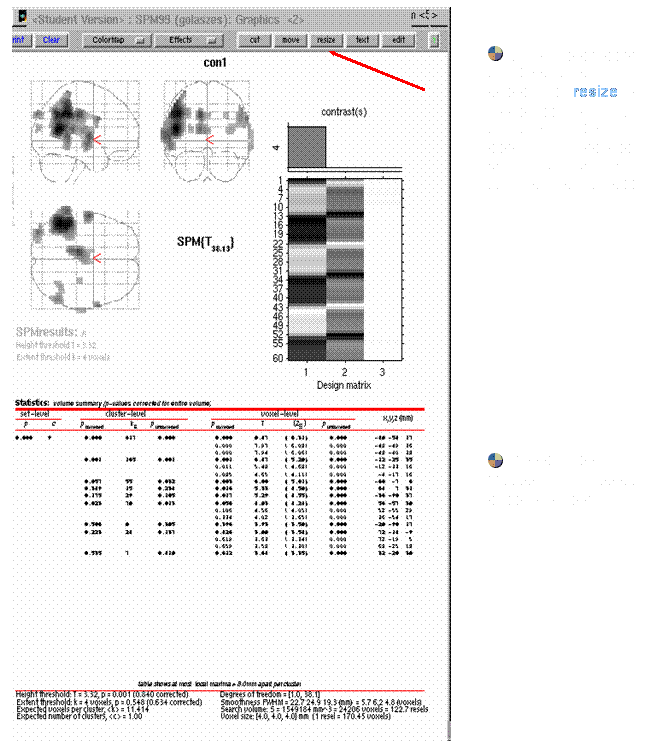
Contrast: in dem
Beispiel wurde ein Trail definiert, dieser wird mit 1
belegt, die Off-Phase mit 0.
Hätte man mit 2 Trails gearbeitet und wollte diese nun von einander
abziehen, so würde man einem Trail die 1 zuweisen und
dem Trail, der davon abgezogen werden soll, eine –1.
Zur Begutachtung der einzelnen Trails würde man 1 0
bzw. 0 1 eingeben (mit Leerzeichen dazwischen!), um Aussagen über die einzelnen
Trails machen zu können.
Fenster
öffnet sich :
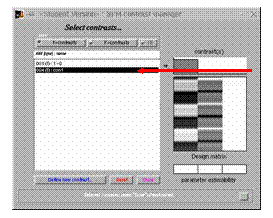
![]() Den
entsprechende Contrast
aus der Liste anwählen
Den
entsprechende Contrast
aus der Liste anwählen ![]() Done anklicken
Done anklicken
Mask with other contrasts : no
title for comparsion
: con1
|
Säule erscheint – warten |
Corrected height treshold : no (no =
uncorrected; yes = corrected)
Treshold (T or p value): 0,001
Extent treshold voxel : 0
Fenster
öffnen sich :
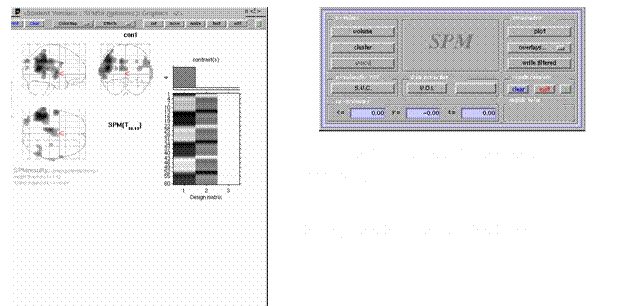
Verschiedene
Möglichkeiten der Darstellung der Ergebnisse
Ø Anzeige der Signifikanzwertetabelle
Volume anklicken
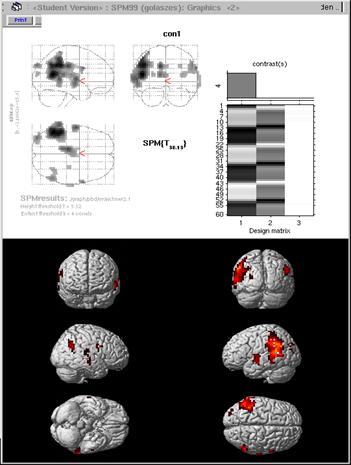
Ø Darstellung
der Ergebnisse auf einem Standart-Gehirn
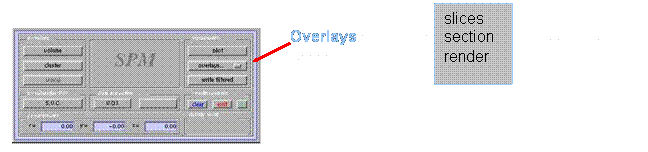
render auswählen Fenster
öffnen sich :
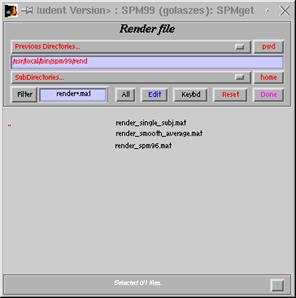
3 Auswahlmöglichkeiten:
1. render_singel_subj.mat
2.
render_smooth_average.mat
3. render_spm96.mat
1.
render_singel_subj.mat :
2 . render_smooth_average.mat :
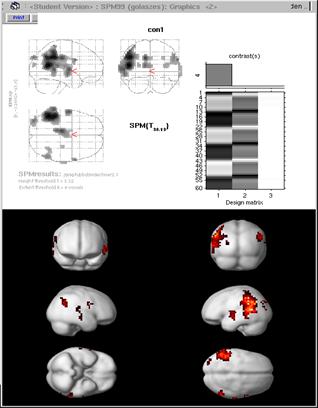
3. render_spm96.mat
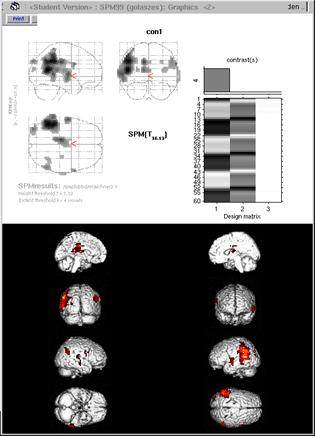
Ø Darstellung der
Ergebnisse auf einem individuellen Gehirn – Schnittbilder
slices auswählen ê: dann den
coregistrierten und normalisierten 3D-Datensatz des
entsprechenden Probanden anklicken,
z.B. 4.-
img
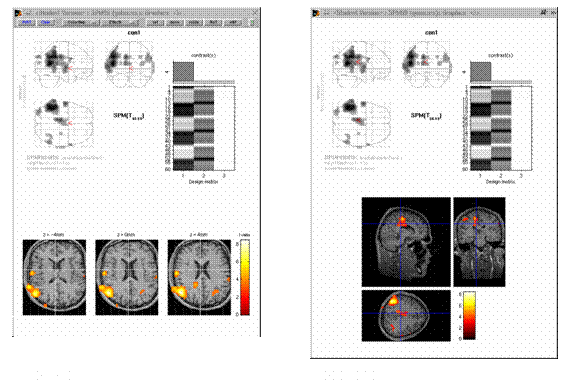
sections auswählen ì : dann
den coregistrierten und normalisierten 3D-Datensatz
des entsprechenden Probanden anklicken, z.B. 4.- img
![]() Durch verschieben
des roten Pfeils im Glasbrain, durch Anklicken mit
der Maus, kann man durch die einzelnen Schnittbilder scrolen.
Ein andere Möglichkeit ist die direkte Eingabe der Koordinaten in das Result-Fenster í.
Durch verschieben
des roten Pfeils im Glasbrain, durch Anklicken mit
der Maus, kann man durch die einzelnen Schnittbilder scrolen.
Ein andere Möglichkeit ist die direkte Eingabe der Koordinaten in das Result-Fenster í.

